AI 신약 개발에서 단백질 유연성이 중요한 이유

AI 신약 개발이란?
제약· 바이오산업에서 기존의 신약 개발은 연구 개발 기간이 오래 걸리고, 천문학적인 비용이 들어가곤 합니다. 식약처 정보를 기준으로 신약을 개발하는 데만 1~2조원의 예산과 10~15년이라는 긴 시간이 소요된다고 합니다. 이러한 한계점을 극복하기 위해 ‘AI 신약 개발’이 등장하게 됐습니다. 현재 AI는 제약뿐만 아니라 정말 다양한 산업에 적용되고 있습니다. AI 신약 개발은 기존 임상 데이터에 AI 기술이 결합하여 신약을 개발하는 것을 의미합니다. 앞으로 신약 개발이라는 산업에 AI가 큰 역할을 수행할 것으로 예측하고 있습니다.
AI 신약 개발 - 단백질 유연성의 중요성
AI 신약 개발 또한 수많은 단계와 과정을 거쳐야 합니다. 약물을 어떻게 디자인할 것인지를 시작으로 표적 파악, 의미 있는 표적과 우수한 화합물을 파악하여 약 후보를 선정하고 사전 임상 시험을 거쳐야 합니다. AI 신약 개발 과정 속에서 모든 과정이 중요하겠지만 오늘 적을 글에서는 ‘단백질의 유연성’에 대해서 적어보려고 합니다. AI 신약 개발을 하는 데 있어서 왜 단백질의 유연성이 중요하고, 언제 사용해야 하는지에 대해 간략하게 설명해드리겠습니다.
단백질의 유연성이란?
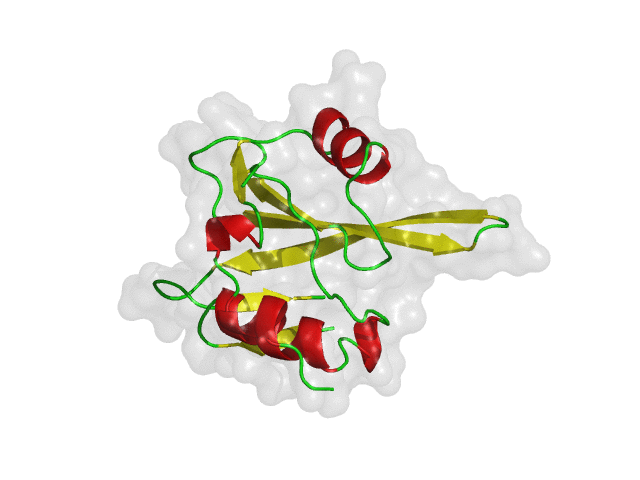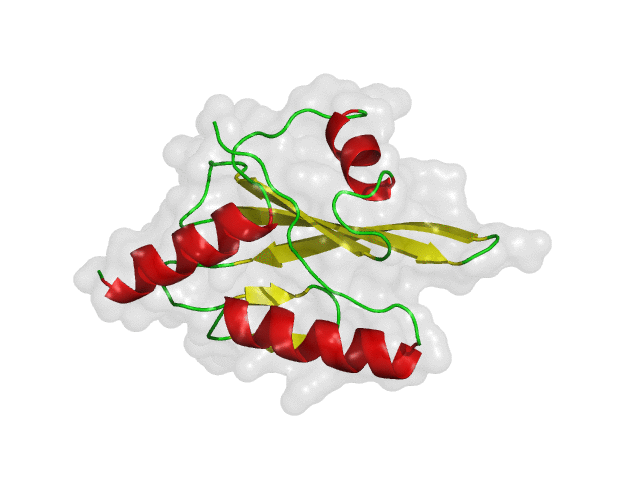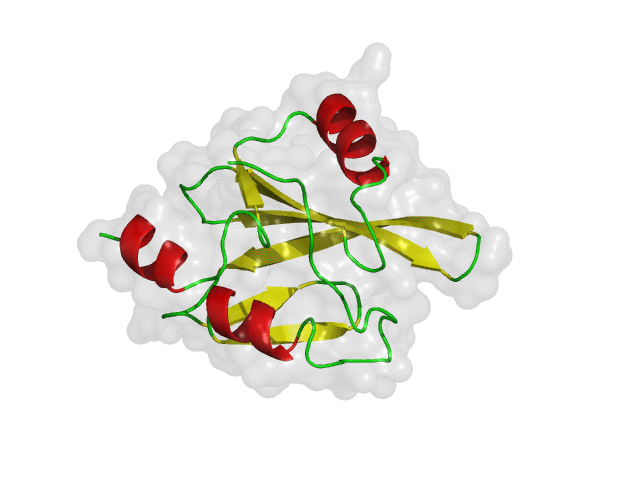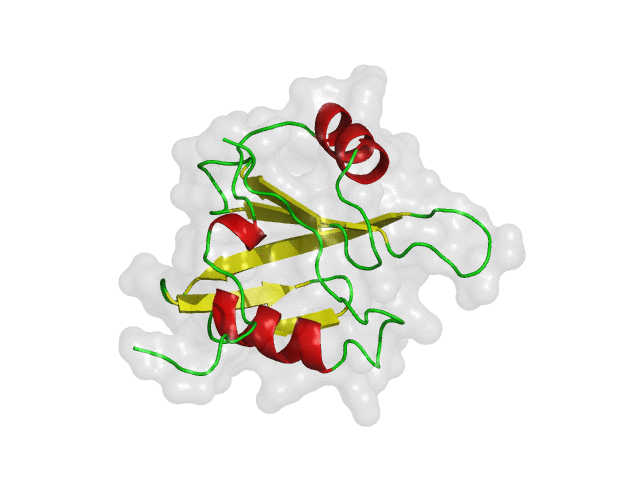
구조 기반 신약 개발(SBDD)의 초기 가상탐색에서 receptor 로 사용되는 단백질은 보통 고정된 구조를 사용하게 됩니다. 이전 글(단백질 구조 선정 방법)에서 소개한 방식을 통하여 한가지의 대표 구조 또는 다양한 약물의 결합 구조를 재현 할 수 있는 몇가지의 단백질 구조를 receptor로 선정하고 가상탐색을 진행하게 됩니다. 하지만 화합물-단백질 결합 구조가 잘 알려지지 않거나 적은 수의 결합 구조를 가지고 있는 경우에는 어려움이 있을 수 있습니다. 또한, 단백질은 생체 내에서 고정되어 있는 구조가 아닌 유연하게 움직이는 것을 생각한다면, 신약 개발 연구에서도 단백질의 유연성을 고려 하는것은 중요한 일입니다.
단백질의 유연성 고려가 필요한 이유
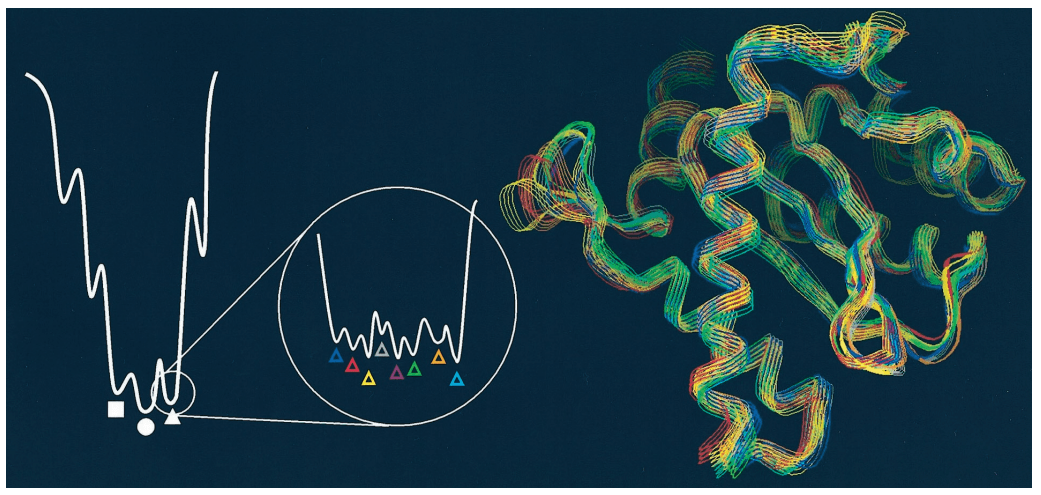
위의 그림은 단백질의 유연성에 따라 다양한 단백질 구조를 가질 수 있다는 것을 간단히 보여주는 예시입니다. 유동적인 단백질은 안정한 에너지 상태를 여러 개 가질 수 있으며 실험적으로 얻어낸 구조는 그중 하나입니다. 따라서, 신약 개발 연구에서 단백질의 유연성을 고려하여 다양한 구조들을 검토 해야 적합한 결합 약물을 찾을 가능성 높일 수 있습니다. 아래의 그림은 다양한 구조를 검토 하였을 때와 그렇지 않았을 때 탐색 할 수 있는 약물의 범위를 보여 줍니다.

단백질의 유연성을 고려한 많은 수의 receptor를 활용하여 가상탐색을 진행한다면 적합한 화합물을 찾을 가능성을 높여주지만, 시간과 비용을 고려한다면 이 또한 적합한 방법은 아닐 것입니다. 탐색할 약물의 수도 많은데, 그에 따라 단백질의 갯수까지 많아진다면 컴퓨터를 사용한 가상탐색일 지라도 그 시간과 비용은 만만치 않을 것입니다.
단백질의 유연성을 고려한 방식들
신약 개발 연구에서 단백질의 유연성을 고려하기 위해 다양한 방법들이 개발 및 활용되고 있습니다. 몇 가지 예를 들어보면, 적합한 약물-단백질 결합 구조를 탐색하기 위한 도킹 방식에서 단백질의 유연성을 고려할 수 있습니다. 또한, 컴퓨터 시뮬레이션을 활용하여 다양하고 변화가 큰 단백질의 구조를 만들어 내어 receptor로 사용하거나, 분자 docking 이후 시뮬레이션을 통해 적합한 약물-단백질의 결합 구조를 찾는 등의 방법이 있습니다.
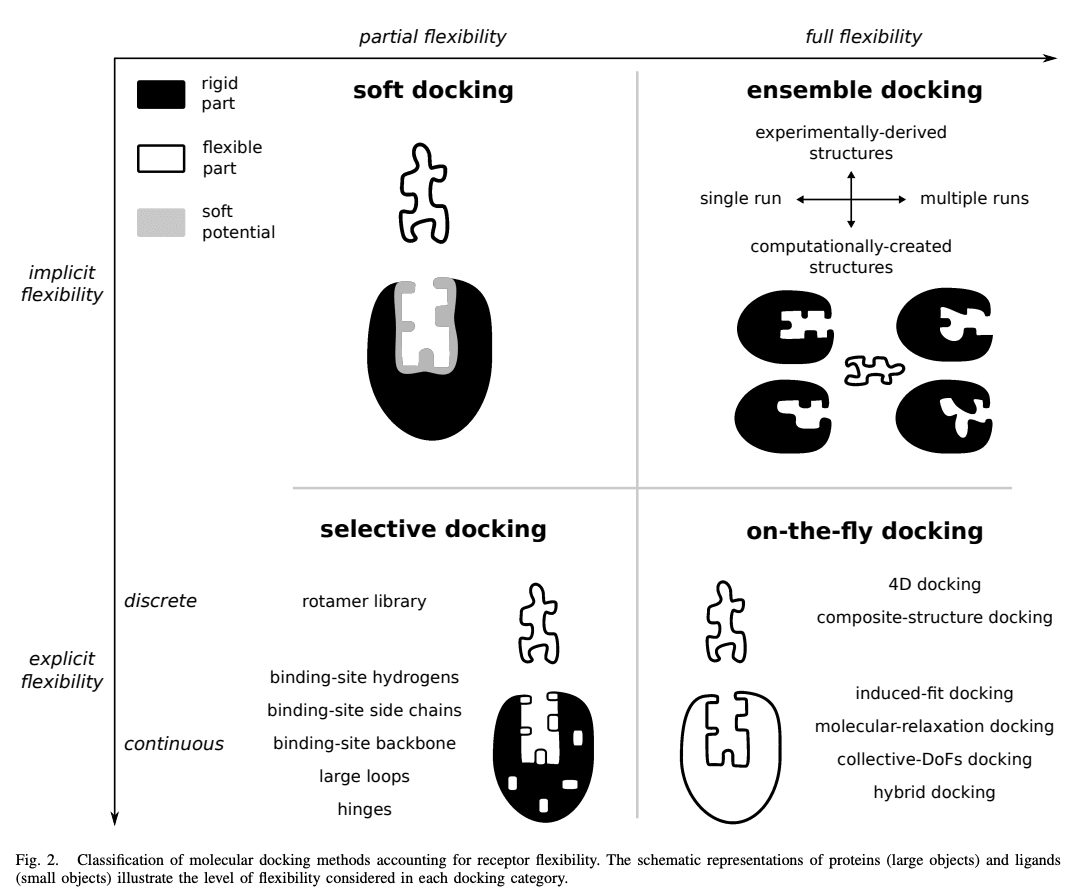
간략히 위의 방식들에 대해 설명하면, 단백질의 결합 위치에서 잔기들의 유연성을 고려하는 soft docking, selective docking 방식, 다양한 단백질 구조를 생성하여 여러 가지 구조를 사용하는 방식(ensemble docking), 그리고 단백질과 화합물을 결합하여 안정화하는 방식(on-the-fly docking) 등이 있습니다.
위에서 이야기하는 방식들에 대해서는 다가오는 포스팅에서 좀 더 자세히 다루어 보기로 하겠습니다.
단백질의 유연성, 언제 활용해야 할까?
실제 신약 개발 연구에서 어떨 때 단백질의 유연성을 고려한 receptor 선정을 해야 하는지에 대해 이야기해 보겠습니다.
1. 결정 구조의 해상도가 안 좋은 경우
먼저, 사용하려는 결정 구조의 해상도(resolution)가 안 좋은 경우입니다. 위에서도 이야기 했듯이 안정한 구조를 갖는 다양한 구조 중 하나의 구조를 실험적으로 얻어내었는데 구조의 신뢰도라고 할 수 있는 해상도가 좋지 않다면 가상탐색에 사용하기에는 여러 가지 문제가 발생 할 수 있습니다. 예를 들면, 약물-단백질 결합구조에서 원자간 충돌이 있다거나, 약물 결합 위치에서 단백질 잔기의 구조가 뒤틀려 있는 경우 등이 있습니다. 이러한 부정확한 정보를 가지고 가상탐색을 진행 한다면 적합한 약물 탐색에 실패할 가능성이 높아집니다. 따라서, 신뢰할 수 있는 안정적인 구조를 갖춘 후 가상탐색에서 사용해야 할 것입니다. 이때 활용할 수 있는 한가지 방식은 분자 동역학 시뮬레이션입니다. 좋지 않은 해상도이지만 실험에 의해 알려진 단백질-약물 결합 결정 구조를 입력 구조로 하고 시뮬레이션을 통해 물리적으로 안정적인 구조를 만들어 내는 것입니다. 이를 통해 원자 간의 충돌, 또는 단백질 잔기의 뒤틀림 등은 해결될 것이고 물리적으로 문제가 발생하지 않는 범위 내의 구조를 갖추게 될 것입니다.

2. 알파폴드 구조를 사용하는 경우
또 다른 경우로는 알파폴드 구조를 사용할 때입니다. 신약 개발 연구에서는 단백질의 결정 구조가 알려지지 않은 단백질을 다루는 경우도 많이 있습니다. 실험적으로 특정 단백질에서 활성이 있는 약물을 알고 있고 해당 단백질에서 결합이 예상되는 잔기 정보까지 알아낸 경우, 해당 단백질에 대한 새로운 약물을 찾거나 약물을 최적화 할 때에 바로 실험을 통해 검증을 하면 좋지만 이는 시간과 비용이 많이 들기 때문에 상대적으로 저렴하고 빠른 컴퓨터를 활용한 연구를 먼저 수행 하게 됩니다. 단백질의 결정 구조가 없이 컴퓨터를 활용한 SBDD 연구를 수행 할 때에 사용되는 단백질 구조로는 가장 정확한 예측 구조로 알려진 알파폴드 구조를 사용하게 됩니다. 하지만 알파폴드 구조는 화합물이 결합되어 있지 않은 apo- 구조가 대부분이기에 가상탐색 및 SBDD 연구를 위한 단백질-화합물 결합 구조를 준비할 필요가 있습니다. 단백질의 유연성을 고려하면 화합물이 결합한 구조가 존재 할 것이라는 것을 실험적으로 검증하여 알고 있기에 이를 토대로 단백질-화합물 결합 구조를 생성 할 수 있습니다. 먼저 알파폴드 구조를 입력 구조를 하여 분자 동역학 시뮬레이션을 수행 합니다. 실험적으로 화합물이 결합할 단백질 잔기를 알고 있기 때문에 결합위치의 변화를 보고 적합한 단백질 구조를 선택 할 수 있습니다. 이어서 해당 약물을 실험들 통해 규명된 단백질 잔기가 있는 결합 위치에 결합한 후 다시 한번 시뮬레이션을 통해 안정화 작업을 수행 하면 가상탐색에 사용 할 단백질-화합물 결합 구조를 얻어 낼 수 있습니다.
이외에도 다양하게 신약 개발 연구에서 단백질의 유연성이 중요시 되는 경우가 있으며, 다가오는 포스팅에서 다양한 방법들과 함께 소개해 드리도록 하겠습니다.
마치며
단백질 유연성을 고려하여 구조를 안정화 할 수 있는 컴퓨터 시뮬레이션 활용 방법도 매우 다양하게 존재하고 있지만 이 또한 시간과 비용이 발생 하는 문제입니다. 적합한 단백질의 접힘 구조를 얻기 위해서는 긴 시간의 시뮬레이션 또는 enhanced sampling 등을 고려한 시뮬레이션 방식의 도입이 필요 할 것입니다. 이러한 방법들에 대해서는 다음 포스팅에서 하나씩 다뤄보겠습니다.